James Lowey est le directeur informatique et le Vice président de TGen, (Translational Genomics Research Institute) ; il nous a expliqué l’importance de l’informatique pour la recherche médicale.
TGen, l’organisme de recherche qui emploie James Lowey, est installé à Phoenix dans le Nevada. Crée par Jeffrey Trente, la référence américaine dans le domaine du génome en 2002, il s’est développé en partie avec des fonds d’états, les dons de sociétés privées qui peuvent déduire de leurs impôts leurs investissements et la vente de ses brevets à l’industrie pharmaceutique. Présent aux journées de l’innovation organisées par Dell, James Lowey présentait les principales évolutions de son groupe et l’implication de Dell dans ses recherches.
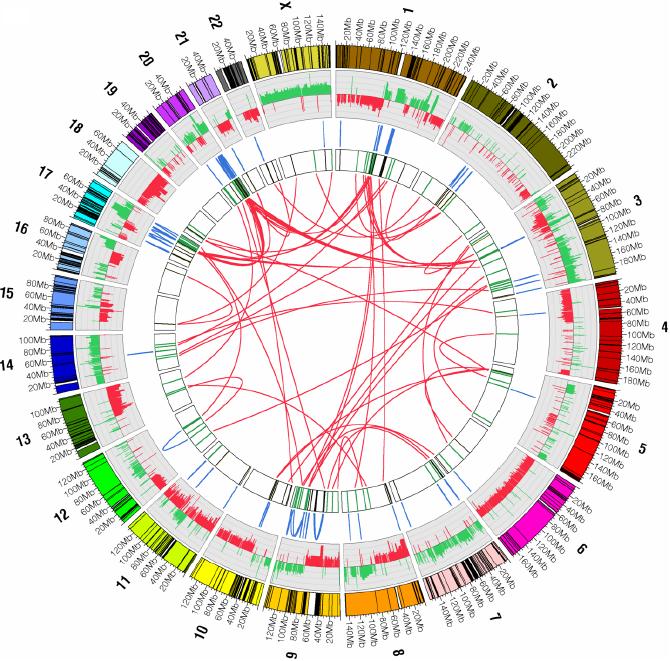
« On est des partenaires de DELL depuis 4 ans, en particulier dans la recherche sur le cancer qui touche les enfants, c’était le désir de Michael Dell. On effectue la recherche sur des dizaines de racks de serveurs de 13 eme génération powerdege (R620 et M420), dont un rack de 10 u avec 512 cœurs Xeon raccordés à des systèmes de stockage Compellent et des réseaux hauts débits. L’objectif, c’est de réduire le temps qu’il faut pour analyser les données moléculaires d’un patient – un processus qui peut prendre de dix jours à six heures. Ces résultats seront désormais répliqués en Europe, en France, et au Liban en particulier. Ce sont une centaine de serveurs multicoeur qui fonctionnent sous linux. L’essentiel des outils employés pour le séquençage du génome humain est d’origine Open Source plus précisément les chercheurs développent en permanence de nouveaux outils pour atteindre les objectifs que l’on s’est fixés. Lorsque je suis arrivé à Tgen je voulais optimiser les premiers programmes de traitement et j’étais arrivé assez rapidement à en faire tourner 35% plus vite.  Mais les chercheurs m’ont dit : cette version de quelques semaines n’est déjà plus utilisée. Difficile de les suivre, c’est une remise en cause permanente. Je me contente désormais avec mon équipe de leurs fournir les meilleurs outils du moment avec la puissance maximale. Je suis en relation avec la plupart des fournisseurs pour obtenir ce qui se fait de mieux. On travaille bien sûr avec tous les systèmes SSD, tous les systèmes in memory et l’on a adopté la solution Fluide cache de Dell qui permet d’optimiser la gestion de tous les disques afin d’obtenir les meilleur temps de réponse. On travaille beaucoup avec les outils Hadoop. On dépend entièrement de la technologie pour progresser. Les docteurs qui font les recherches ont une image pour représenter le travail qu’ils effectuent sur le génome. C’est un peu comme un puzzle de 3 milliards de pièces qui serait pris dans une tornade puis éparpillé dans un champ. C’est à eux de reconstituer le puzzle le plus rapidement possible. Avec l’aide de nos sponsors on a pu réaliser des recherches en France et au Liban qui n’étaient possibles qu’aux USA. »
Mais les chercheurs m’ont dit : cette version de quelques semaines n’est déjà plus utilisée. Difficile de les suivre, c’est une remise en cause permanente. Je me contente désormais avec mon équipe de leurs fournir les meilleurs outils du moment avec la puissance maximale. Je suis en relation avec la plupart des fournisseurs pour obtenir ce qui se fait de mieux. On travaille bien sûr avec tous les systèmes SSD, tous les systèmes in memory et l’on a adopté la solution Fluide cache de Dell qui permet d’optimiser la gestion de tous les disques afin d’obtenir les meilleur temps de réponse. On travaille beaucoup avec les outils Hadoop. On dépend entièrement de la technologie pour progresser. Les docteurs qui font les recherches ont une image pour représenter le travail qu’ils effectuent sur le génome. C’est un peu comme un puzzle de 3 milliards de pièces qui serait pris dans une tornade puis éparpillé dans un champ. C’est à eux de reconstituer le puzzle le plus rapidement possible. Avec l’aide de nos sponsors on a pu réaliser des recherches en France et au Liban qui n’étaient possibles qu’aux USA. »
InformatiqueNews : « En quoi l’évolution des études sur le séquençage du génome est plus rapide que celle de l’informatique ?
James Lowey : « Pendant des années, on a comparé la loi de Moore à celle de l’évolution de la génomique. Dan le cas de Moore, tous les deux ans, la puissance d’un serveur est multipliée par deux pour un tarif équivalent. Et bien, si l’on compare le séquençage du génome, les prix ont baissé beaucoup depuis depuis dix ans. En 2003, année de la découverte, séquencer les 3 milliards de paires de bases de l’ADN humain, le prix d’un sequencage coûtait 3, 8 milliards maintenant on est à près de 1000 dollars.
On est en train de construire dans une de nos filiales Ashion Analytics, une Appliance qui permettra de séquencer rapidement votre ADN. Dans un proche futur, si vous souffrez d’un cancer de l’estomac, le système sera en mesure de préconiser les médicaments pour le soigner au mieux. Un séquenceur crée des rapports qui facilitera les traitements ».
InformatiqueNews : « Que représente en terme de volumes un séquençage et sur quels volumes de données travaillez-vous ?
James Lowey : « Dans les domaines du génome, on a des formats de données standards très utilisés qui exploitent des fichiers de 10, 50 ou 10 Go mais nous dans la recherche, on traite souvent des fichiers de 1 téraoctet et souvent des fichiers de 4To. C’est l’un des enjeux de cette recherche, manipuler des volumes de données importants avec une grande vélocité. On a beaucoup évolué depuis avec des formats type qui se sont allongés aux fur et à mesure des découvertes. »
InformatiqueNews : « Avez-vous crée une sorte de langage spécifique aux données et utilisez-vous le cloud ?
James Lowey : « On ne travaille pas beaucoup sur le cloud public, même si l’on dit souvent que notre outil principal, c’est un navigateur. Amazon web service propose des applications sur le séquençage et des exemples pour se familiariser. Mais dans notre cas nos recherches doivent déboucher sur la dépose de brevets que l’on désire vendre pour financer notre fonctionnement. Cela nécessite des précautions. On partage bien sûr un certain nombre d’outils mais ce n’est pas l’essentiel. Mais on utilise le cloud pour certaines opérations quand à la création d’un langage spécifique aux données du génome, c’est l’un de nos objectifs « .
Le mode d’intervention de Tgen en France et au Liban ne nous a pas été détaillé mais l’on peut supposer qu’il s’agira d’un appui informatique sur le séquençage à des équipes existantes. La France est l’un des pays les plus actifs dans le monde pour la recherche sur le génome. La recherche française uniquement sur les cancers mobilise environ 6 000 chercheurs, enseignants et techniciens en France, soit plus de 700 équipes.






 puis
puis