Le numérique envahit tous les secteurs économiques et toutes les activités humaines. La santé est un grand bénéficiaire, notamment dans le domaine de l’imagerie médicale.
Que de progrès depuis le fameux stéthoscope de Laennec découvert dans le milieu du 19e siècle ! C’était là les premiers vers une auscultation de l’intérieur du corps humain. Puis furent découverts les rayons X qui permirent d’avoir les premières images du corps humain. La puissance de calcul des ordinateurs depuis une cinquantaine d’années ont permis de faire des progrès considérables permettant de décoder des données liées à des vibrations acoustiques ou des ondes électromagnétiques et à les transformer en informations exploitables et facilitant le diagnostic. En utilisant des algorithmes et des données de références, l’ordinateur a permis de passer de l’âge du décodage passif à celui d’aide à l’interprétation et au diagnostic.
Aujourd’hui, les images médicales sont omniprésentes dans la pratique médicale courante et hospitalière. Outre les radiographies, quatre grandes modalités d’imagerie sont couramment utilisées : le scanner, l’IRM, l’échographie, ou la scintigraphie (1). Les images produites par ces quatre modalités sont volumiques : elles fournissent en chaque point du corps humain des informations mesurées dans un petit élément de volume appelé voxel, l’extension volumique du pixel.
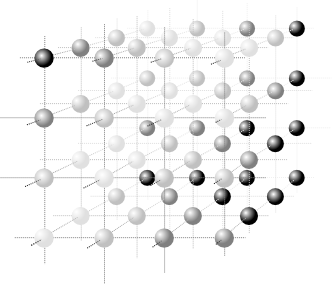 Schématisation d’une matrice de voxels en nuances de gris
Schématisation d’une matrice de voxels en nuances de gris
Il existe d’autres modalités d’imagerie du corps humain, et de nouvelles techniques émergent régulièrement. Par exemple l’élastographie (2) qui permet de mesurer l’élasticité des tissus à partir d’IRM ou d’ultrasons, et l’endomicroscopie qui permet de visualiser l’architecture microscopique des cellules à l’extrémité de fibres optiques.
 Ces différents sujets seront traités par Nicholas Ayache dans une série de leçons portant sur l’imagerie médicale computationnelle dans le cadre de sa chaire annuelle « Informatique et sciences numériques » au Collège de France. A cette occasion, il présentera certaines des recherches les plus avancées en imagerie médicale computationnelle dans le cadre de son cours « Le patient numérique personnalisé : images, médecine, informatique ». La leçon inaugurale qui s’est tenue le 10 avril a présenté comment l’informatique et les sciences numériques permettent d’augmenter le contenu des images médicales pour construire un modèle numérique et personnalisé du patient, au service de la médecine et de la chirurgie modernes (Pour écouter la leçon inaugurale). Les principes algorithmiques, mathématiques et biophysiques de cette jeune discipline – le traitement informatique des images médicales – ont été illustrés par des applications cliniques variées.
Ces différents sujets seront traités par Nicholas Ayache dans une série de leçons portant sur l’imagerie médicale computationnelle dans le cadre de sa chaire annuelle « Informatique et sciences numériques » au Collège de France. A cette occasion, il présentera certaines des recherches les plus avancées en imagerie médicale computationnelle dans le cadre de son cours « Le patient numérique personnalisé : images, médecine, informatique ». La leçon inaugurale qui s’est tenue le 10 avril a présenté comment l’informatique et les sciences numériques permettent d’augmenter le contenu des images médicales pour construire un modèle numérique et personnalisé du patient, au service de la médecine et de la chirurgie modernes (Pour écouter la leçon inaugurale). Les principes algorithmiques, mathématiques et biophysiques de cette jeune discipline – le traitement informatique des images médicales – ont été illustrés par des applications cliniques variées.
Ce jeune champ de recherche, à la croisée de l’informatique, des sciences numériques et de la médecine, a pour objectif de concevoir et développer des logiciels de traitement informatique des images médicales pour assister le médecin dans sa pratique clinique. Ces logiciels visent notamment à enrichir le diagnostic en extrayant, à partir d’images médicales, des informations objectives et cliniquement utiles. Ils visent également à assister la pratique thérapeutique avec des algorithmes de planification et de simulation appliqués à un modèle numérique du patient.
Des volumes de données considérables
La plupart des images médicales sont très volumineuses. L’image anatomique d’un organe, voire du corps entier peut contenir entre quelques millions et plusieurs centaines de millions de voxels, stockés dans d’immenses matrices 3-D de nombres (à la différence de ce que l’on appelle le big data et dont la source et la nature des données est très hétérogène). La quantité d’information augmente rapidement lorsque plusieurs images sont acquises sur un même patient pour exploiter la complémentarité des différentes modalités, ou pour suivre une évolution temporelle ; il s’agit alors d’images 4-D avec trois dimensions spatiales et une dimension temporelle.
Le rôle de l’informatique et des sciences numériques
Face à toutes ces images et à leur complexité, le médecin ne peut souvent extraire visuellement que des informations lacunaires et qualitatives. Les images volumiques ne sont souvent visualisées que sous la forme de coupes 2-D. Il est alors quasiment impossible de quantifier précisément le volume d’une tumeur, de détecter une anomalie isolée dans un organe entier et suivre son évolution subtile entre deux examens, ou de quantifier dans une série temporelle d’images le mouvement d’un organe dynamique comme le cœur. Il est encore plus difficile de planifier une intervention délicate sans l’aide de l’ordinateur.
L’informatique et les sciences numériques jouent alors un rôle crucial pour exploiter de façon rigoureuse et optimale cette surabondance d’information. Elles sont essentielles pour l’analyse des images reconstruites dont le but est d’extraire de façon objective l’information cliniquement pertinente et de la présenter dans un cadre unifié et intuitif au médecin. Elles offrent également la possibilité de construire un modèle numérique du patient pour la simulation : simulation de l’évolution d’une pathologie ou de l’effet d’une thérapie par exemple, ou simulation de gestes médicaux ou chirurgicaux pour l’entrainement du praticien.
Analyse et simulation informatiques des images médicales reposent sur des algorithmes qui doivent prendre en compte la spécificité de l’anatomie et de la physiologie humaines à l’aide de modèles mathématiques, biologiques, physiques ou chimiques, adaptés à la résolution des images. Ces modèles du corps humain dépendent eux-mêmes de paramètres permettant de modifier la forme et la fonction des organes simulés. Utilisés avec un jeu de paramètres standard, les modèles sont génériques : ils décrivent et simulent la forme et la fonction moyennes des organes dans une population. Mais avec les images médicales et l’ensemble des données disponibles sur un patient spécifique, les paramètres d’un modèle générique peuvent être ajustés grâce à des algorithmes pour reproduire plus précisément la forme et la fonction des organes de cet individu. On dispose alors d’un modèle personnalisé.
Patient numérique personnalisé et médecine computationnelle
Le patient numérique personnalisé (3) n’est autre que cet ensemble de données numériques et d’algorithmes permettant de reproduire à diverses échelles la forme et la fonction dynamique des principaux tissus et organes d’un patient donné. C’est aussi le cadre unifié qui permet d’intégrer les informations provenant des images anatomiques et fonctionnelles du patient, ainsi que les informations qui décrivent l’histoire singulière du patient et de sa maladie.
Les modèles numériques et personnalisés du patient sont destinés à assister le médecin dans sa pratique médicale : assister le diagnostic en quantifiant l’information présente dans les images ; assister le pronostic en simulant l’évolution d’une pathologie ; assister la thérapie en planifiant, simulant et contrôlant une intervention. Voilà ce qui préfigure la médecine computationnelle de demain, une composante informatique de la médecine qui n’a pas vocation à se substituer au médecin, mais qui est destinée à lui fournir des outils numériques pour l’assister dans l’exercice de sa pratique médicale au service du patient.
Des images médicales au patient numérique
Dans sa leçon inaugurale, intitulée « des images médicales au patient numérique », Nicholas Ayache choisi quatre exemples qui illustrent une certaine progression des algorithmes et des modèles mis en œuvre pour exploiter les images médicales. Les deux premiers exemples, morphométrie et endomicroscopie computationnelles, relèvent du domaine de l’anatomie computationnelle. Les algorithmes utilisés s’appuient sur des modèles géométriques, statistiques et sémantiques du corps humain. Les deux exemples suivants, oncologie et cardiologie computationnelles, relèvent de la physiologie computationnelle. Leurs algorithmes s’appuient en plus sur des modèles biologiques, physiques ou chimiques du corps humain.
Conclusion
L’imagerie médicale computationnelle, à la croisée de l’informatique et de l’imagerie médicale, fournit de nouveaux outils numériques au service du médecin et du patient, dans le cadre plus large de la médecine computationnelle.
Les progrès actuels dans ces domaines permettent d’entrevoir comment l’informatique et les sciences numériques peuvent accompagner le passage d’une médecine normalisée et réactive à une médecine plus personnalisée, préventive et prédictive (4). Ils reposent en grande partie sur des avancées algorithmiques en traitement d’images et dans la modélisation numérique de l’anatomie et de la physiologie du corps humain.
| Les quatre mousquetaires de l’imagerie médicale avancée (source : Wikipédia)Scanner La tomodensitométrie (TDM), dite aussi scanographie, tomographie axiale calculée par ordinateur (TACO), CT-scan (CT : computed tomography), CAT-scan (CAT : computer-assisted tomography), ou simplement scanner pour l’appareil est une technique d’imagerie médicale qui consiste à mesurer l’absorption des rayons X par les tissus puis, par traitement informatique, à numériser et enfin reconstruire des images 2D ou 3D des structures anatomiques.IRM L’imagerie par résonance magnétique (IRM) est une technique d’imagerie médicale permettant d’obtenir des vues 2D ou 3D de l’intérieur du corps de façon non invasive avec une résolution en contraste relativement élevée. L’IRM repose sur le principe de la résonance magnétique nucléaire (RMN) qui utilise les propriétés quantiques des noyaux atomiques pour la spectroscopie en analyse chimique.Echographie L’échographie est une technique d’imagerie employant des ultrasons. L’électronique de l’échographe se charge d’amplifier et de traiter ces signaux afin de les convertir en signal vidéo. L’image se fait en niveaux de gris selon l’intensité de l’écho en retour. Scintigraphie |
__________
1. Scanner ou tomodensitométrie par rayons X ; IRM ou imagerie par résonance magnétique ; échographie ou imagerie ultrasonore ; scintigraphie : TEP (tomographie par émission de positons) et TEMP (tomographie d’émission monophotonique).
2. M. Fink, Renversement du temps, ondes et innovation ; leçon inaugurale, Collège de France, 2009.
3. On utilise aussi l’expression de patient virtuel, notamment dans le cadre de la simulation d’interventions médicales et chirurgicales. Bien entendu, il ne faut pas confondre patient virtuel et Malade imaginaire.
4. Elias Zerhouni, Grandes tendances de l’innovation biomédicale au XXIe siècle ; leçon inaugurale, Collège de France, 2011.
Les leçons de Nicholas Ayache au Collège de France
29 avril 2014 16:30 – 17:30
Sciences des images médicales : les grandes classes de problèmes
06 mai 2014 16:30 – 17:30
Se repérer dans les images : recalage et segmentation
13 mai 2014 16:30 – 17:30
Variabilité anatomique et fonctionnelle : atlas statistiques
20 mai 2014 16:30 – 17:30
La dimension temporelle : quantifier une évolution
27 mai 2014 16:30 – 17:30
Imagerie des tumeurs : modèles biophysiques pour mesurer et prédire
03 juin 2014 16:30 – 17:30
Imagerie microscopique in vivo : mosaïques numériques et indexation
10 juin 2014 16:30 – 17:30
Le coeur numérique personnalisé : diagnostic, pronostic et thérapie
17 juin 2014 16:30 – 17:30
Réalité virtuelle, simulation, et perspectives





 puis
puis